Die Einzelzell-Transkriptom-Analysen, die noch vor kurzer Zeit eine futuristische Vision waren, haben unser Verständnis der zellulären Heterogenität revolutioniert und uns neue Zelltypen in allen Organismen finden lassen. Durch die Entwicklung von Einzelzell-Sequenziermethoden ist es heute möglich zelluläre Entwicklungswege oder Krankheitsverläufe in Tiefe besser zu verstehen. Für alle Einzelzellanalysen müssen jedoch die Zellen aus dem Gewebeverbund gelöst werden, so dass uns die räumliche Information der Zelltypen und damit das Verständnis der zellulären Mikroumgebung, der interzellulären Kommunikation oder Funktion des Gewebes verloren gehen.
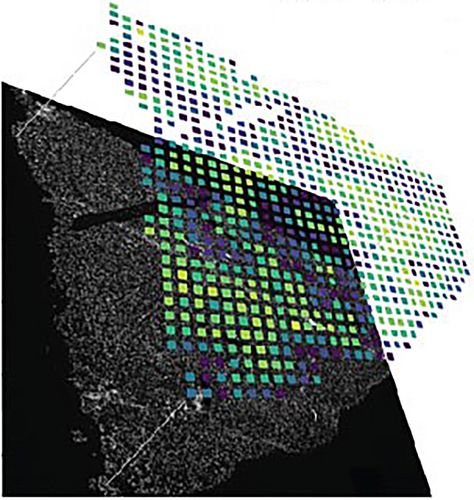
Um räumlich aufgelöste Einzelzell-Transkriptom-Daten zu gewinnen, müssen Bildgebungs- und Sequenzierungstechniken in Kombination arbeiten. Mehrere Ansätze wurden in den letzten Jahren entwickelt, um die Zusammenführung von Bild- und Einzelzell-sequenzierdaten zu vereinigen. Dabei werden je nach Forschungsfrage verschiedene Parameter wie räumliche Auflösung, Nachweisgrenze, Screening-Bereich, Zugänglichkeit, Kompatibilität mit bestehenden Arbeitsabläufen und Kosten gegeneinander abgewogen. Ein früheres Analyseverfahren machte sich die Idee zu nutze, die örtliche Information mittels eines DNA-Barcodes an das Zielmolekül zu hängen. Nach Extraktion aller Zielmoleküle und anschließendem massenhaften Sequenzieren kann das Barcodesignal benutzt werden, um ein künstliches Bild zu generieren. Eines der größten Probleme dieses Verfahrens war der geringe Durchsatz und gleichzeitig die hohen Kosten für die Analyse.
Einen Ausweg aus diesem Dilemma war die Entwicklung eines verbilligten Multiplexingverfahrens. Dabei hat Johannes Wirth, Doktorand im Labor von Matthias Meier, einen fortschrittlichen Arbeitsablauf entwickelt, der eine Multiplexing-Methode für örtlich aufgelöste Transkiptomdaten gepaart mit hochwertiger Bildgebung (genannt xDBiT) bietet und damit die zuvor berichteten Sequenzieransätze erheblich weiterentwickelt. Im Zentrum stand die Entwicklung eines neues mikrofluidischen Chip, der es ermöglicht, Zielmoleküle in großflächigen Gewebeabschnitten kostengünstig zu codieren. Im Vergleich zur ursprünglichen Methode konnte im neuen Ansatz die Anzahl der Bildinformationen pro Bildpunkt um das 6- bzw. 12-fache erhöht werden. Umgekehrt bedeutet dies, dass wir in der Lage sind ca. 5000 Gene pro Bildpunkt aufzulösen - das ermöglichte zum Beispiel seltene Zelltypen wie Podozyten in den Glomeruli der Niere aufzulösen. Zusätzlich zu den technischen Fortschritten stellte das Team auch eine Open-Source-Analysepipeline zur Verfügung, um die Methode leicht zugänglich zu machen.
Die entwickelte Analysemethode ermöglicht Multiplexing, erhöht die Sequenzierungstiefe, verbessert die Qualität der Bilddaten und bietet damit ein robustes und genaues Analysewerkzeug für räumliche Genexpressionsmuster. Da das Verfahren für eine Vielzahl von Geweben geeignet ist, wird es Studien zu komplexen Krankheiten und Multiorgan(dys-)funktionen erleichtern.
Originalpublikation in Nature Communications:
Spatial transcriptomics using multiplexed deterministic barcoding in tissue
DOI: 10.1038/s41467-023-37111-w
Zur Pressemitteilung der Universität Leipzig: